Biochemische Regulation
CDPKs (engl. calcium-dependent protein kinases) zeichnen sich dadurch aus, dass intramolekular die Bindung von Kalzium über vier EF-Hand Motive in der Kalzium-bindenden Sensordomäne unmittelbar über eine Konformationsveränderung die Aktivierung der Protein-Kinase Effektordomäne vermittelt. Zusätzlich tritt Enzymregulation über Phosphorylierung, katalyisert durch die CDPK selbst (Stimulus-induzierte Autophosphorylierung) oder durch weitere Protein-Kinasen sowie Dephosphorylierung durch Protein-Phosphatasen auf.
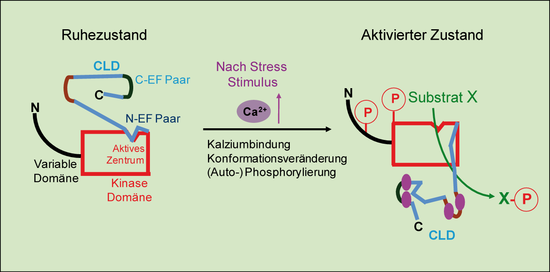
Schematisches Modell zur in vivo Aktivierung von CDPKs. CDPKs bilden eine Genfamilie von Serin- und Threonin Protein-Kinasen. Die Mitglieder verfügen über eine konservierte Struktur aus einer N-terminalen variablen Domäne, einer Protein-Kinase Effektordomäne, einer autoinhibitorischen Region und einer C-terminalen „Calmodulin-like“ Domäne (CLD), welche meist vier konservierte EF-Hand Motive zur Kalziumbindung aufweist. Das Modell berücksichtigt neben eigenen experimentellen Ergebnissen auch die CDPK Kristallstruktur des Protisten Toxoplasma gondii (Wernimont et al., 2010).
Wir untersuchen, wie die verschiedenen Kalzium-bindenden EF-Hand Motive bzw. N- und C-terminaler EF-Hand Paare sowie Phosphorylierungsmuster die Konformation und Aktivität des Enzyms in vivo kontrollieren und damit die funktionale Diversifikation einzelner CDPK Mitglieder in den jeweiligen Signalwegen bestimmen. Für das Arabidopsis Enzyme AtCPK21 konnte von uns ein unterschiedlicher Beitrag der einzelnen Kalzium-bindenden EF-Hand-Motive auf die Kalzium-abhängige Kinase-Aktivität aufgezeigt werden (Franz et al., 2011).