Maßgeschneiderte Liganden für G-Protein gekoppelte Rezeptoren
GPCRs als Signalmaschinerie
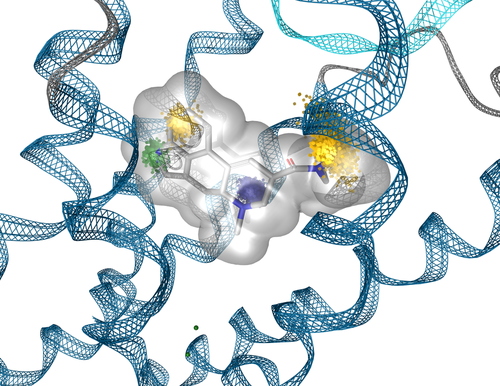
Dynophore von LSD im Serotonin-Rezeptor erklärt funktionelle Selektivität (biased Signaling)
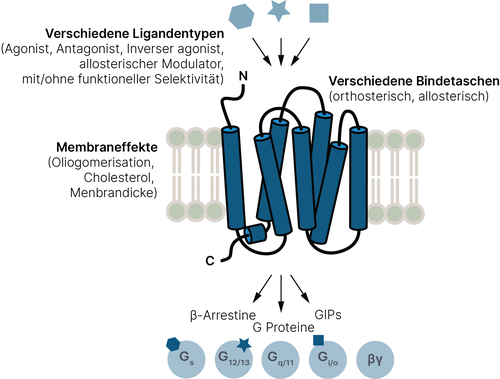
G-Protein-gekoppelte Rezeptoren (GPCRs) sind eine integrative und hochdynamische Signalmaschinerie, die chemisch codierte Informationen über mehrere Signalwege durch die Zellmembran übertragen (Bioorg. Med. Chem., 23:3907-3912, 2015, Trends Pharmacol. Sci., 35:630-638, 2014). Ca. 700 Arzneistoffe (ca. 35 % aller zugelassenen) wirken direkt auf 134 bekannte GPCRs (Molecular Pharmacology, 93, 251-258, 2018) in einem breiten Indikationsspektrum. Trotz dieser hohen biologischen und pharmakologischen Relevanz sind die zugrunde liegenden Mechanismen in Bezug auf die Rezeptordynamik weitestgehend unverstanden, was auf die komplexe Natur der GPCR-Signalübertragung zurückzuführen ist (Chem Rev, 117, 4-20, 2014). Viele bereits am Markt befindliche partielle GPCR-Agonisten (z. B. Pilocarpin zur Glaukomtherapie oder Buprenorphin als Analgetikum) wurden nicht rational entwickelt, d. h., der Mechanismus der partiellen Aktivierung ist mechanistisch nicht verstanden.
Mit den Arbeiten von Brian Kobilka (Nobelpreis 2016) zur Kristallisation der β‑Adrenozeptoren startete eine Revolution in der Strukturbiologie, durch die heute (Stand: Januar 2023) 498 Kristallstrukturen von GPCRs verfügbar sind und die diese Rezeptorklasse dem strukturbasierten, rationalen Wirkstoffdesign zugänglich machte. Die Rezeptordynamik und das damit verbundene Downstream-Signaling bleiben aber eine große Herausforderung, da Kristallstrukturen niemals das Konformationsensemble des Rezeptors abbilden können. Erst durch Simulationen kann die für die biologische Wirkung relevante Rezeptordynamik abgeschätzt werden. Ein mechanistisches Verständnis der ligandenabhängigen Rezeptoraktivierung ist essenziell für das rationale Design von GPCR-Modulatoren, die mit einem für die pharmakologische Wirkung essenziellen Signalingprofil verknüpft sind.
Unsere Arbeitsgruppe hat sich in den letzten Jahren darauf spezialisiert, Modelle zu entwickeln, die GPCRs als dynamische Einheiten betrachten und ihr komplexes Signalrepertoire berücksichtigen. Ein wichtiges Ziel ist es, die spezifische Aktivierung eines bestimmten Weges (funktionelle Selektivität/Bias) zu adressieren, um maßgeschneiderte Liganden für die Aktivierung eines bestimmten intrazellulären Pathways zu entwickeln. Das Alleinstellungsmerkmal dieser Modelle ist eine dynamische Betrachtung des Rezeptors unter besonderer Berücksichtigung dynamischer Ligandenbindemuster. Dazu haben wir eine neue Modellierungstechnik entwickelt, dynamische 3D Pharmakophore, die die Vorteile des klassischen strukturbasierten Designs mit der Komplexität einer Molekulardynamiksimulation verbindet.
Das langfristige Ziel ist die Entwicklung von Liganden mit maßgeschneiderter funktioneller Selektivität. Als Modellsysteme fokussieren wir uns vorerst auf Muskarinrezeptoren und Opioidrezeptoren (Mol. Inf. 34:526-530, 2015, Bioorg. Med. Chem. Lett., 26:4769-4774, 2016, J. Med. Chem. 57:6739-6750, 2014, Molecules, 25:2087, 2020, Molecules, 26:3267, 2021, Pharmaceuticals, 15:680, 2022), da für diese Rezeptoren ein hohes therapeutisches Potenzial und ausreichende Daten verfügbar sind. In Kooperation mit Prof. Dr. Holzgrabe (Würzburg) und Prof. Dr. Mohr/Prof. Dr. Weindl (Bonn) konnten wir auf atomistisch-mechanischer Ebene zeigen, wie allosterische Reste bitopischer Liganden das G-Protein-Rekrutierungsprofil und somit das intrazelluläre Signaling verändern (ACS Chem. Biol., 12:1743-1748, 2017, ACS Pharmacol. Transl. Sci., 3:859-867, 2020). Unabhängig von diesem Effekt konnten wir am M2-Rezeptor die Existenz von Ligandenbindungsensembles nachweisen, bei denen ein Ligand zwei unterschiedliche Bindungsmodi einnehmen kann, wobei einer den aktiven und der andere den inaktiven Rezeptorzustand stabilisiert (J. Biol. Chem., 291:16375-16389, 2016).
Maßgeschneiderte funktionelle Selektivität an Opioidrezeptoren kann dosislimitierende Nebenwirkungen wie Atemstillstand (wie z. B. für das 2020 in den USA zugelassene Oliceridin (µOR) berichtet wurde) verringern. Für die Entwicklung sicherer Analgetika durch Liganden mit funktioneller Selektivität des κ‑Opioid-Rezeptors läuft aktuell bis 2024 ein DFG-Projekt. In einer aktuellen Arbeit (Molecules, 28:718, 2023) konnten wir ein mechanistisches Modell des Diterpenoids Salvinorin A aus Salvia divinorum am κ‑Opioid-Rezeptor entwickeln, das als Basis für neuartige, ungeladene Modulatoren dienen wird.
Wir bedanken und für finanzielle Unterstützung dieses Forschungsbereichs durch: