Neue computergestützte Methoden im Wirkstoffdesign
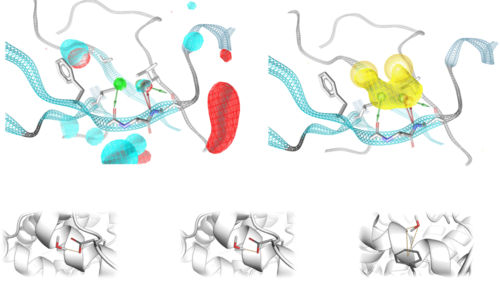
Pyrod nutzt die schnelle Kinetik der Wassermoleküle in der Molekulardynamik zur Ableitung von Interaktionsmuster für virtuelles Screening
Bildquelle: G. Wolber / D. Schaller
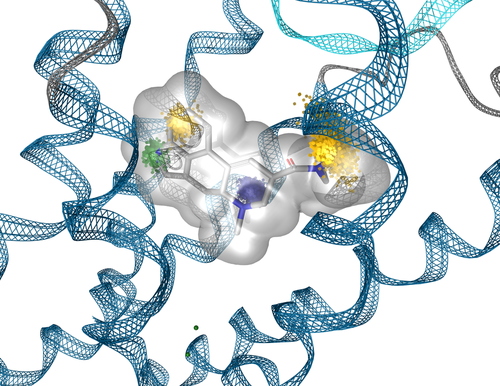
Dynophore von LSD im Serotonin-Rezeptor erklärt funktionelle Selektivität (biased Signaling)
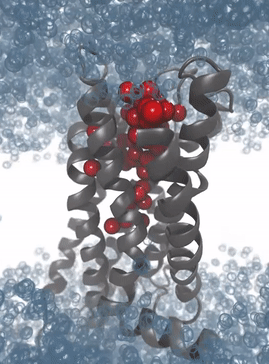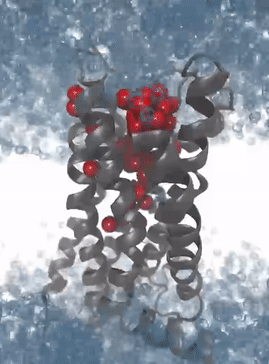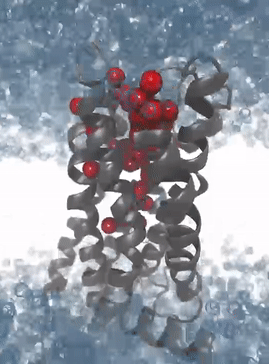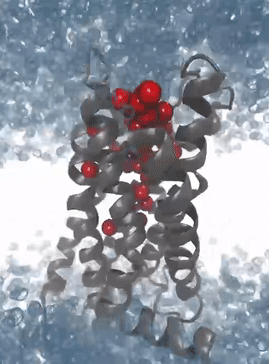
Simulation der Proteindynamik des M2-Rezeptors: Die Solvatisierungsmuster der Bindetasche ermöglichen das Erkennen von Interaktionsmustern zur Entwicklung von 3D Pharmakophoren für virtuelles Hochdurchsatzscreening
Mit immer leistungsfähigeren Computern und der Verfügbarkeit von General Purpose Computation on Graphics Processing Units (GPGPUs, Berechnungen auf Grafikkarten) wurden Molekulardynamiksimulationen zur Routine im pharmazeutischen Wirkstoffdesign. Die von uns kürzlich entwickelten Programme Dynophores (dynamische 3D Pharmakophormodelle) und PyRod erlauben die systematische Analyse der großen Datenmengen aus Molekulardynamiksimulationen und deren Verwendung für das Wirkstoffdesign und für virtuelles Screening. Insbesondere bei Liganden von G-Protein-gekoppelten Rezeptoren stellt die Verwendung von Dynophores ein wichtiges Werkzeug dar, um kleine Unterschiede in der Ligandenbindung mit der damit verbundenen Konformationsänderung des ternären Komplexes und dem damit assoziierten intrazellulären Signaling zu verstehen und zu modellieren. Die Analyse aller verfügbaren Daten für eine bestimmte Fragestellung (‚Big Data‘) und maschinelles Lernen sind dabei integraler Bestandteil aller Modeling-Ansätze in unserer Arbeitsgruppe.
In der pharmazeutisch-chemischen Forschung steht für das Entwickeln neuer Wirkstoffe, deren Charakterisierung, Synthese und Optimierung primär die Anwendung computergestützter Methoden im Mittelpunkt. Im klassischen strukturbasierten Wirkstoffdesign konnte meine Arbeitsgruppe in der Anwendung von virtuellem Screening, 3D Pharmakophoren, Docking, Ensemble-Docking und Molekulardynamiksimulationen langjährige Erfahrung sammeln. Neue Methoden der künstlichen Intelligenz unterstützen unsere Syntheseideen vor allem im De-novo-Design. Durch structure-informed learning kombinieren wir die Vorteile von maschinellem Lernen mit der Transparenz von strukturbasiertem Design, was vor allem für das Verständnis ligandeninduzierter Konformationsänderungen für Rezeptoren wichtig ist.
Aktuelle Dynophore-Anwendungen:
- M. Janežič, K. Valjavec, K. B. Loboda, B. Herlah, I. Ogris, M. Kozorog, M. Podobnik, S. G. Grdadolnik, G. Wolber, and A. Perdih. Dynophore-based approach in virtual screening: a case of human DNA Topoisomerase IIα, Int J Mol Sci, 22(24):13474, 2021. [doi:10.3390/ijms222413474]
- P. Durairaj, L. Fan, D. Machalz, G. Wolber, and M. Bureik. Functional characterization and mechanistic modeling of the human cytochrome P450 enzyme CYP4A22, Febs Lett, 593(16):2214-2225, 2019. [doi:10.1002/1873-3468.13489]
- J. Mortier, J. R. C. Prévost, D. Sydow, S. Teuchert, C. Omieczynski, M. Bermudez, R. Frédérick, and G. Wolber. Arginase structure and inhibition: Catalytic site plasticity reveals new modulation possibilities, Scientific Reports, 7(1):13616, 2017. Links: [doi:10.1038/s41598-017-13366-4]
- B. Nizami, D. Sydow, G. Wolber, and B. Honarparvar. Molecular insight on the binding of NNRTI to K103N mutated HIV-1 RT: molecular dynamics simulations and dynamic pharmacophore analysis, Mol Biosyst, 12:3385-3395, 2016. [doi:10.1039/C6MB00428H]
- A. Bock, M. Bermudez, F. Krebs, C. Matera, B. Chirinda, D. Sydow, C. Dallanoce, U. Holzgrabe, M. De Amici, M. J. Lohse, G. Wolber, and K. Mohr. Ligand Binding Ensembles Determine Graded Agonist Efficacies at a G Protein-Coupled Receptor, J Biol Chem, 291(31):16375-16389, 2016. [doi:10.1074/jbc.M116.735431]
Aktuelle PyRod-Anwendungen:
- S. Pach, T. M. Sarter, R. Yousef, D. Schaller, S. Bergemann, C. Arkona, J. Rademann, C. Nitsche, and G. Wolber. Catching a moving target: comparative modeling of flaviviral NS2B-NS3 reveals small molecule Zika protease inhibitors, ACS Medicinal Chemistry Letters, 11(4):514-520, 2020. [doi:10.1021/acsmedchemlett.9b00629]
- D. Schaller, and G. Wolber. Pyrod enables rational homology model-based virtual screening against MCHR1, Mol Inf, 39(6):e2000020, 2020. [doi:10.1002/minf.202000020]
Übersichtsartikel:
- Noonan, K. Denzinger, V. Talagayev, Y. Chen, K. Puls, C. A. Wolf, S. Liu, T. N. Nguyen, and G. Wolber. Mind the gap - deciphering GPCR pharmacology using 3D pharmacophores and artificial intelligence, Pharmaceuticals, 15(11):1304, 2022.[doi:10.3390/ph15111304]
- D. Schaller, D. Sribar, T. Noonan, L. H. Deng, T. N. Nguyen, S. Pach, D. Machalz, M. Bermudez, and G. Wolber. Next generation 3D pharmacophore modeling, WIREs Comp. Mol. Sci., 10(4):e1468, 2020. [doi: 10.1002/wcms.1468]